山东数据统计生命科学软件下载
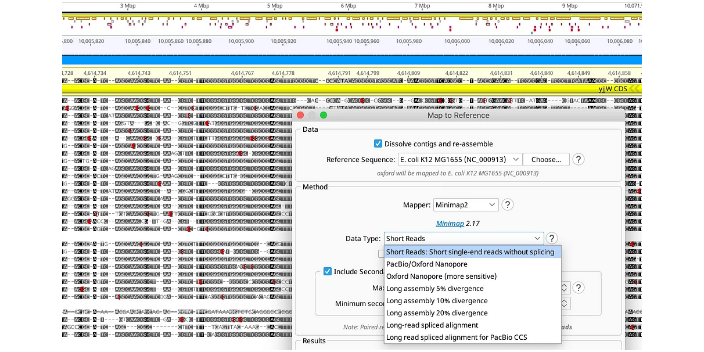
载体构建——同源重组法-生命科学软件。还是以基因ATG7为例,首先在NCBI,TAIR等基因网站找到这个基因,复制CDS序列。将序列插入newdnafile,然后重命名文件。选中全长后,点击Features>addfeature>labelname改为CDS>type改为CDS。这样的目的是为了方便后续检测基因是否插入成功。查看实验室已有的酶,点击Emazy>chooseemazy>先移除右边框中的酶,然后再添加你所在实验室中已购买的酶。保存文件为ATG文件。后续要在载体中插入到这个文件所以要先保存。-生命科学软件。打开载体文件,找到到多酶切位点序列。查看哪些酶切位点可用且不会切到基因片段,然后确定酶切位点,可用一个酶,也可以用两个酶,不过比较好选两个酶。在载体文件中选择要两个酶切位点,点击Action>In-fusioncloning>Insertfragment。生命科学领域所需要的哪些软件。山东数据统计生命科学软件下载
基于SnapGene软件的多序列比对教程-生命科学软件打开SnapGene,直接将txt拖入snapgene起始界面。SnapGene将自动识别txt中的每个序列,并将其拆分成为单独的序列文件,点击“Import”,软件将生成一个文件夹,文件夹中含有txt中的每一个FASTA序列。-生命科学软件用SnapGene打开任意一个序列(推荐打开文件大小比较大的),选择Tools菜单栏中的“AlignMultipleSequences”功能(快捷键Ctrl+L)在弹出的窗口中,将剩下几个序列都选中,点击“打开”,SnapGene将对选中的序列进行多重比对福建FCS Express生命科学软件哪家好生命科学软件有什么特点和功能分析。
特征-生命科学软件。DNA可视化查看DNA序列的多个视图地图:自定义,酶位点,特征,引物,ORF,DNA颜色等的显示。地图可以是圆形或线性格式。序列:使用双链模式查看酶位点,具有翻译,引物和DNA颜色的特征。单链模式显示具有彩色特征的紧凑概览。酶:从一系列酶组中选择。剪切网站可以显示为数字或行,按名称或频率排序。-生命科学软件。特征:按名称、位置、大小、颜色,方向性或类型对带注释的要素列表进行排序。复选框在紧凑或完全展开的显示之间的切换。
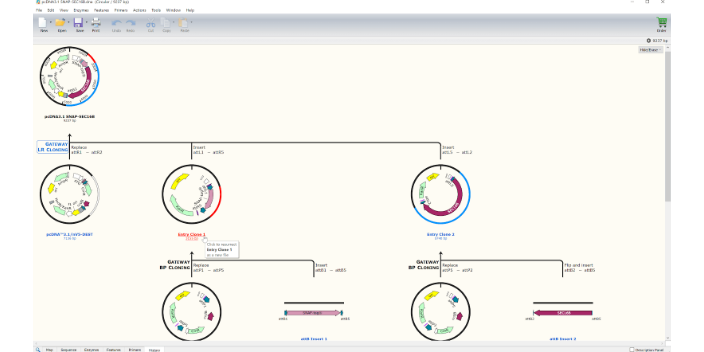
Gibson组装-生命科学软件。通过融合多达八个片段或通过将多达八个片段插入向量来模拟GibsonAssembly。GibsonAssembly是一种流行的无缝克隆方法。选择要融合的片段及其方向,SnapGene将设计引物。线性化的载体可以通过酶消化或反向PCR产生。IN-FUSION®克隆-生命科学软件。通过将八个片段插入载体来模拟Clontech的In-Fusion克隆。融合克隆是创建无缝基因融合的一种非常通用的方法。选择要融合的片段及其方向,SnapGene将设计引物。线性化的载体可以通过酶消化或反方向PCR产生。有没有什么学生物必备(推荐)的软件?
历史记录颜色-生命科学软件。使用可选的历史记录标上颜色来标识序列的***更改。查看有关组装结构的详细信息,包括结扎的粘性末端。拥有您的数据SnapGene可以帮助您选择读取和共享文件,同时保持对数据的完全控制。安全文件管理-生命科学软件。把您的SnapGene文件放在您想要的地方。使用您熟悉的、安全的计算机操作系统来存储和管理SnapGene文件。从其他格式导入支持读取许多常见的文件格式。不仅可以导入DNA序列,还可以导入功能注释和注解。医学生物行业常用专业软件介绍。山东数据统计生命科学软件下载
生命科学软件有什么特点和功能。山东数据统计生命科学软件下载
将序列粘贴到序列框中,同理更改名字以及添加酶切位点序列(下游酶切位点是BamHI)和保护碱基的序列,然后点击“addprimertotemplate”-生命科学软件点击“Map”,Ctrl键选择两个引物(如图),点击“Action”→“PCR”点击“PCR”,即获得扩增产物-生命科学软件打开表达载体序列,按Ctrl键选择两个酶切位点(蓝色标记的),点击“Action”→“Restrictioncloning”→“Insertfragment”点击“Insert”,选择刚刚扩增产物的文件“A”,然后分别输入相应内切酶的名字,点击插入的片段。在右下角点击“Clone”即可。山东数据统计生命科学软件下载
上一篇: 湖南正版生命科学软件多少钱
下一篇: 湖北图表制作生命科学软件试用